NVIDIA NGC팀은 실시간 Q&A로 진행되는 웨비나를 개최하고 NGC 카탈로그의 주피터 노트북(Jupyter notebook)을 심층 분석합니다. 이 리소스들로 여러분의 AI 여정을 시작하는 방법을 알아보십시오. ‘NVIDIA NGC 주피터 노트북 데이: 의료 이미지 세그멘테이션’에 지금 등록하세요.
이미지 세그멘테이션(image segmentation)은 이미지가 보다 유의미하고 분석하기 쉽게 표현되도록 변경함으로써 하나의 디지털 이미지를 다수의 세그먼트로 분할합니다. 의료 이미지 분야에서 이미지 세그멘테이션은 장기와 이상 상태를 식별, 측정, 분류하며 더 나아가 진단 관련 정보의 발견에도 기여합니다. 이 과정에는 X선과 자기공명영상(MRI), 컴퓨터단층촬영(CT), 양전자단층촬영(PET) 등의 다양한 형식으로 수집한 데이터가 사용됩니다.
각각의 활용 사례가 추구하는 정확도와 성능을 갖춘 최첨단 모델을 구축하려면 적합한 환경을 설정하고, 최적의 하이퍼파라미터로 훈련을 진행하며, 원하는 정확도를 달성할 수 있도록 최적화해야 합니다. 이 모든 과정에는 상당한 시간이 소요되는데요. 그래서 데이터 과학자와 개발자에게는 지난한 작업들을 신속히 해결하기에 적합한 툴셋이 필요합니다. 이를 위해 개발된 것이 바로 NGC 카탈로그입니다.
NGC 카탈로그는 GPU 최적화 AI와 HPC 애플리케이션, 각종 툴의 허브입니다. NGC를 사용하면 성능 최적화 컨테이너에 간편하게 접근하고 사전 훈련된 모델로 모델 개발 시간을 단축하는 한편, 산업별 SDK로 완벽한 AI 솔루션을 구축하고 AI 워크플로우를 가속할 수 있습니다. 이처럼 다양한 에셋은 컴퓨터 비전과 음성 인식, 언어 이해에 이르는 여러 활용 사례에 사용됩니다. NGC를 기반으로 자동차, 헬스케어, 제조, 소매 등의 여러 산업에서 구축될 각종 솔루션에도 기대가 모아지고 있습니다.
U-Net을 활용한 3D 의료 이미지 세그멘테이션
이번 포스팅에서는 의료 3D 이미지 세그멘테이션 노트북(Medical 3D Image Segmentation notebook)으로 MRI 이미지에서 뇌종양을 예측하는 방법을 소개합니다. AI 입문자와 이미지 세그멘테이션 기법의 의료 이미지 적용에 특별한 관심을 가진 누구나 본 포스팅을 참고하실 수 있습니다. 3D U-Net은 3D 볼륨의 원활한 세그멘테이션을 수행하면서 고도의 정확도와 성능을 보장합니다. 또한 세그멘테이션과 관련한 다양한 문제의 해결에 사용되기도 합니다.
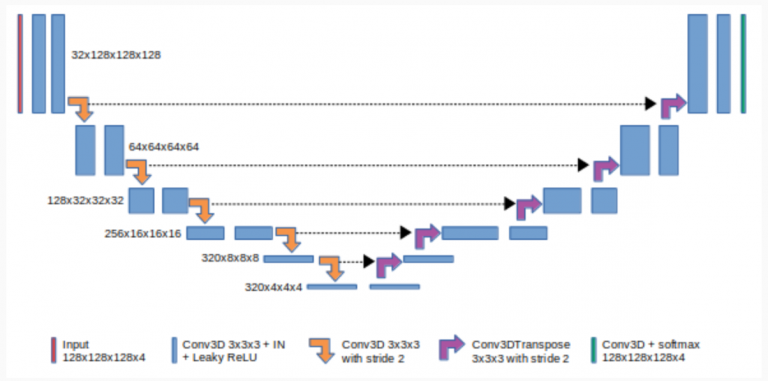
Figure 2는 3D U-Net이 수축 경로(좌측)와 확장 경로(우측)로 구성되어 있음을 보여줍니다. 패딩(padding)이 없는 합성곱(convolutions) 연산을 반복 적용하고 맥스 풀링(max pooling)으로 다운샘플링을 진행합니다.
딥 러닝에서 합성곱 신경망(CNN)은 심층 신경망의 하위 개념으로, 주로 이미지의 인식과 처리에 사용됩니다. CNN은 딥 러닝을 통해 생성(generative) 작업과 기술(descriptive) 작업 모두를 수행하며 머신 비전(machine vision)과 추천 시스템, 자연어 처리 등에 이용됩니다.
CNN에서 패딩은 특정 CNN의 커널이 이미지를 처리할 때 추가되는 픽셀의 수를 뜻합니다. 패딩이 없는 CNN은 해당 이미지에 추가된 픽셀이 없다는 의미입니다.
풀링은 CNN에서 다운샘플링을 진행하는 방식입니다. 맥스 풀링은 대표적인 풀링 기법의 하나로 특정 피처의 가장 활성화된 부분을 요약해 보여줍니다. 확장 경로의 모든 단계는 피처맵(feature map)의 업샘플링(upsampling)과 해당 부분을 축소 경로에서 잘라낸 피처 맵의 연결(concatenation)로 구성됩니다.
요구 사항
이 리소스에는 TensorFlow NGC 컨테이너를 확장하고 종속성 일부를 캡슐화하는 도커파일(Dockerfile)이 포함됩니다. 다음의 명령을 사용하여 리소스를 다운로드할 수 있습니다.
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/unet3d_medical_for_tensorflow/versions/20.06.0/zip -O unet3d_medical_for_tensorflow_20.06.0.zip
이 종속성과 더불어 다음의 구성 요소도 필요합니다.
- NVIDIA Docker
- NGC의 최신 TensorFlow 컨테이너
- NVIDIA Ampere 아키텍처, NVIDIA Turing, 또는 NVIDIA Volta GPU
혼합 정밀도(mixed precision)나 TF32 정밀도로 Tensor Core를 사용하는 모델, 또는 FP32를 사용하는 모델을 훈련하려면, 뇌종양 세그멘테이션(Brain Tumor Segmentation) 2019 데이터세트 기반 3D U-Net 모델의 기본 파라미터를 활용하여 다음의 과정을 수행하세요.
리소스 다운로드
리소스 페이지의 우측 상단에 있는 점 세 개를 클릭하여 리소스를 수동으로 다운로드합니다.
또한 다음의 wget 명령을 사용할 수도 있습니다.
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/unet3d_medical_for_tensorflow/versions/20.06.0/zip -O unet3d_medical_for_tensorflow_20.06.0.zip
U-Net TensorFlow NGC 컨테이너 구축
이 명령은 Docker파일을 사용하여 unet3d_tf라는 명칭의 Docker 이미지를 생성하고, 필요한 구성 요소 일체를 자동으로 다운로드합니다.
docker build -t unet3d_tf .
데이터세트 다운로드
뇌종양 세그멘테이션 2019 데이터세트 웹사이트에 사용자로 등록하고 데이터를 확보하세요. 다운로드한 데이터는 컨테이너의 /data가 마운트된 곳에 위치해야 합니다.
컨테이너 실행
NGC 컨테이너에서 대화형 세션을 시작하여 전처리와 훈련, 추론을 진행하려면 다음의 명령을 실행해야 합니다. 이 명령은 컨테이너를 시작하고 ./data 디렉터리를 볼륨 형태로 컨테이너 내 /data 디렉터리에 마운트하며 ./results 디렉터리를 컨테이너의 /results 디렉터리에 마운트합니다.
컨테이너의 장점은 필수 라이브러리 일체와 종속성을 단일의 독립된 환경에 패키징할 수 있다는 점입니다. 이렇게 하면 복잡한 설치 프로세스에 대해 걱정할 필요가 없습니다.
mkdir data
mkdir results
docker run --runtime=nvidia -it --shm-size=1g --ulimit memlock=-1 --ulimit stack=67108864 --rm --ipc=host -v ${PWD}/data:/data -v ${PWD}/results:/results -p 8888:8888 unet3d_tf:latest /bin/bash
컨테이너 내에서 노트북 시작하기
다음의 명령을 사용하여 컨테이너 내에서 주피터 노트북(Jupyter notebook)을 시작하세요.
jupyter notebook --ip 0.0.0.0 --port 8888 --allow-root
데이터세트를 컨테이너 내 /data 디렉터리로 옮기세요. 다음의 명령으로 해당 노트북을 다운로드합니다.
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/med_3dunet/versions/1/zip -O med_3dunet_1.zip
다운로드한 노트북을 주피터랩(JupyterLab)에 업로드하고 노트북의 셀을 실행하여 데이터세트의 전처리와 모델의 훈련, 벤치마크, 검증을 진행합니다.
다음 블로그에서 이어집니다!