주피터 노트북
주피터 노트북의 셀을 실행하면 사전에 다운로드한 데이터세트를 확인하고 뇌종양 이미지들을 볼 수 있습니다. 확인을 마친 후 데이터 전처리 명령을 참고하여 훈련용 데이터를 준비합니다. 다음 단계는 모델을 훈련하고, 해당 훈련 과정의 체크포인트를 활용하여 예측을 수행하는 것입니다. 마지막으로 예측 함수의 출력값을 시각적으로 확인합니다.
이미지 확인
데이터세트의 확인에는 니바벨(nibabel)을 사용합니다. 니바벨은 대표적인 의료, 신경 이미지 파일 형식에 대한 읽기와 쓰기 기능을 제공하는 패키지입니다.
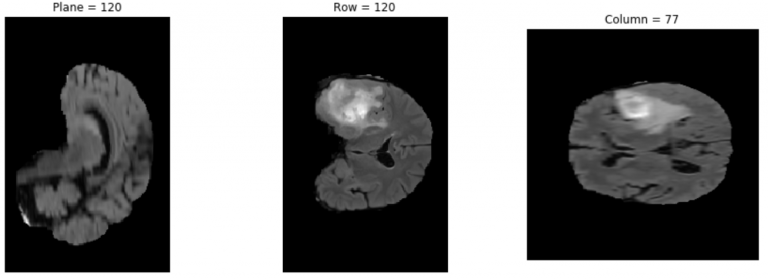
다음의 세 가지 셀을 실행하면 pip 인스톨로 니바벨을 설치하고, 데이터세트에서 이미지를 선택한 다음, 매트플롯라이브러리(matplotlib)로 데이터세트에서 골라낸 제3의 이미지를 플로팅(plotting)할 수 있습니다. 코드의 이미지 주소를 변경하여 다른 데이터세트의 이미지들도 확인이 가능합니다.
import nibabel as nib
import matplotlib.pyplot as plt
img_arr = nib.load('/data/MICCAI_BraTS_2019_Data_Training/HGG/BraTS19_2013_10_1/BraTS19_2013_10_1_flair.nii.gz').get_data()
def show_plane(ax, plane, cmap="gray", title=None):
ax.imshow(plane, cmap=cmap)
ax.axis("off")
if title:
ax.set_title(title)
(n_plane, n_row, n_col) = img_arr.shape
_, (a, b, c) = plt.subplots(ncols=3, figsize=(15, 5))
show_plane(a, img_arr[n_plane // 2], title=f'Plane = {n_plane // 2}')
show_plane(b, img_arr[:, n_row // 2, :], title=f'Row = {n_row // 2}')
show_plane(c, img_arr[:, :, n_col // 2], title=f'Column = {n_col // 2}')
결과는 Figure 4와 유사한 형태로 얻어집니다.
데이터 전처리
dataset/preprocess_data.py 스크립트는 원시 데이터를 훈련과 평가에 사용되는 TFRecord 형식으로 변환합니다. 2019 BraTS 챌린지(2019 BraTS challenge)로 구축한 데이터세트에는 교모세포종(GBM/HGG)과 중증도가 낮은 신경교종(LGG)의 MRI 스캔이 3TB 이상 포함되어 있습니다. 이들은 다중의 기관에서 정기 검사를 통해 임상적으로 확보한 수술 전, 다중모달 스캔이며 병리학적 진단 또한 완료된 것들입니다. 경우에 따라서는 환자의 전체 생존 기간(OS) 관련 데이터도 포함돼 있습니다. 이 데이터는 훈련, 인증, 검증 데이터세트로 구성됩니다.
이미지 형식은 nii.gz.입니다. NIfTI는 신경 이미지용 파일 형식입니다. 다운로드한 데이터세트를 전처리하려면 다음의 명령을 실행하세요.
python dataset/preprocess_data.py -i /data/MICCAI_BraTS_2019_Data_Training -o /data/preprocessed -v처리를 마친 이미지의 최종 형식은 tfrecord입니다. 데이터를 효율적으로 읽을 수 있도록 데이터를 직렬화(serialize)하고, 각각이 선형적으로 읽힐 수 있는 파일 세트(각각 100MB~200MB)에 저장합니다. 이는 해당 데이터가 네트워크를 통해 스트리밍되는 경우에 특히 중요합니다. TFRecord는 바이너리 레코드(binary records) 시퀀스를 저장하는 간단한 형식으로, 데이터 로딩 프로세스의 속도를 현저히 높입니다.
기본 파라미터로 훈련 진행하기
도커 컨테이너를 구성한 후 기본 하이퍼파라미터로 싱글 폴드(폴드 0)의 훈련을 시작할 수 있습니다(예를 들어 {1 to 8} GPUs {TF-AMP/FP32/TF32}).
Bash examples/unet3d_train_single{_TF-AMP}.sh <number/of/gpus> <path/to/dataset> <path/to/checkpoint> <batch/size>
가령 단일 GPU에서 32비트 정밀도(FP32 또는 TF32)와 배치 사이즈 2로 훈련을 진행하려면 다음의 명령을 수행하세요.
bash examples/unet3d_train_single.sh 1 /data/preprocessed /results 2GPU당 배치 사이즈를 2로 설정한 GPU 8개와 혼합 정밀도(TF-AMP)로 싱글 폴드를 훈련하려면 다음의 명령을 수행합니다.
bash examples/unet3d_train_single_TF-AMP.sh 8 /data/preprocessed /results 2훈련 성과 벤치마킹
훈련의 성과는 벤치마킹 스크립트를 실행하여 평가할 수 있습니다.
bash examples/unet3d_{train,infer}_benchmark{_TF-AMP}.sh <number/of/gpus/for/training> <path/to/dataset> <path/to/checkpoint> <batch/size>
이 스크립트는 모델을 실행하고 성과를 보고합니다. 예를 들어 GPU 4개에서 배치 사이즈2와 TF-AMP로 훈련한 결과를 벤치마크하려면 다음의 명령을 실행하세요.
bash examples/unet3d_train_benchmark_TF-AMP.sh 4 /data/preprocessed /results 2예측
검증용 데이터세트를 사용하고 exec 모드로 예측을 진행하여 모델을 검증할 수 있습니다. 결과는 model_dir 디렉터리에 저장되며, 해당 데이터세트의 경로는 data_dir입니다.
python main.py --model_dir /results --exec_mode predict --data_dir /data/preprocessed_test예측 결과 플로팅
다음의 코드 예제에서는 \results 폴더에서 선택한 결과의 하나를 플로팅합니다.
import numpy as np
from mpl_toolkits import mplot3d
import matplotlib.pyplot as plt
data= np.load('/results/vol_0.npy')
def show_plane(ax, plane, cmap="gray", title=None):
ax.imshow(plane, cmap=cmap)
ax.axis("off")
if title:
ax.set_title(title)
(n_plane, n_row, n_col) = data.shape
_, (a, b, c) = plt.subplots(ncols=3, figsize=(15, 5))
show_plane(a, data[n_plane // 2], title=f'Plane = {n_plane // 2}')
show_plane(b, data[:, n_row // 2, :], title=f'Row = {n_row // 2}')
show_plane(c, data[:, :, n_col // 2], title=f'Column = {n_col // 2}')
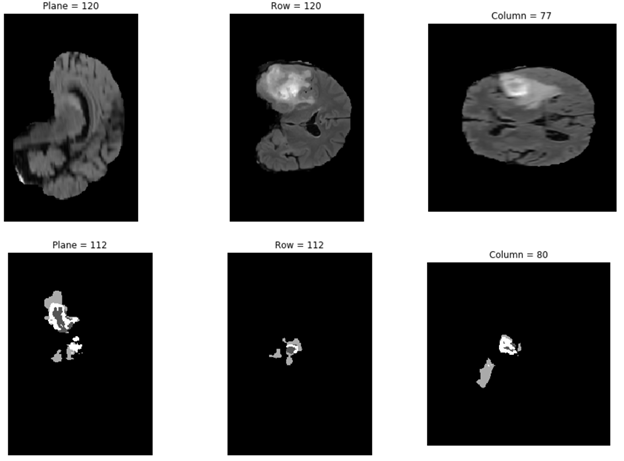
고급 옵션
이 노트북에 내장된 고급 기능을 탐색하려면 -h 또는 –help를 사용해 main.py 옵션들의 전체 목록을 살펴보세요. 다음의 셀을 실행하면 이 스크립트의 실행 모드와 기타 파라미터를 변경하는 방법을 확인할 수 있습니다. 이 스크립트를 사용하여 커스터마이징된 하이퍼파라미터로 모델의 훈련, 예측, 평가, 추론을 진행할 수 있습니다.
python main.py --helpmain.py 파라미터는 훈련, 평가, 예측 등 다양한 작업을 수행하도록 변경할 수 있습니다.
기본 하이퍼파라미터를 사용하여 해당 모델을 훈련할 수도 있습니다. python main.py –help 명령을 실행하면, 훈련용 하이퍼파라미터를 비롯하여 여러분이 변경할 수 있는 인수 목록의 확인이 가능합니다. 예를 들어 훈련 모드에서는 학습률을 기본값 0.0002에서 0.001로, 훈련 단계를 16000에서 1000으로 변경할 수 있습니다. 이를 위해서는 다음의 명령을 수행하세요.
python main.py --model_dir /results --exec_mode train --data_dir /data/preprocessed_test --learning_rate 0.001 --max_steps 1000
main.py의 기타 실행 모드도 수행할 수 있습니다. 일례로 이번 포스팅에서는 다음의 명령을 수행하여 python.py의 예측 실행 모드를 사용했습니다.
python main.py --model_dir /results --exec_mode predict --data_dir /data/preprocessed_test결론
이번 포스팅에서는 NGC 카탈로그의 간단한 주피터 노트북을 사용하여 의료 이미지 모델의 구축을 시작하는 방법을 살펴봤습니다. 이 주피터 노트북을 응용하여 자신만의 고유한 의료 이미지 워크플로우를 구축할 때 NVIDIA Clara Train을 사용할 수 있습니다. Clara Train에는 AI가 지원하는 주석화(Annotation) API와 주석화 서버가 포함되어 있으며, 이들은 의료용 뷰어 일체와 원활히 통합되어 AI의 활용을 지원합니다. 이 훈련용 프레임워크에는 여러분의 AI 워크플로우를 위한 연합 학습과 전이 학습 등 분산식 학습 기법들이 포함됩니다.
이러한 리소스를 사용하여 여러분의 AI 여정을 시작하는 방법을 배우려면 실시간 Q&A로 진행되는 NVIDIA NGC 주피터 노트북 데이: 의료 이미지 세그멘테이션 웨비나에 지금 등록하세요!