다른 몇몇 선행 연구들에 이어, 인간 뇌 프로젝트(Human Brain Project, HBP)는 뇌 연구, 인지신경과학, 기타 뇌 관련 과학을 증진 시키고자 하는 포부를 갖고 있습니다. 유럽위원회(EC)에서 2013년에 창안된 HBP는 뇌와 뇌질환 관련 데이터를 수집, 정리, 배포하고 뇌 자체를 시뮬레이션 하는 것을 목표로 하고 있는데요¹
이러한 연구 활동을 진행하기 위해 HBP가 필요로 하는 것은 좋은 지도입니다. 약 1천억개의 뉴런과 100조 개의 연결망으로 이루어져 있는 인간의 뇌는 현재까지 인류에게 알려져 있는 것 중 가장 복잡한 시스템이기 때문이지요.
이처럼 복잡하기 짝이 없는 뇌 지도를 매핑하기 위해 독일 율리히 연구소(Forschungszentrum Jülich)의 연구진들은 인간 뇌의 3D 멀티모달(multi-modal) 모델을 개발하고 있습니다. 연구진은 현미경과 첨단 이미지 분석 기법으로 수천 개의 아주 얇은 뇌 세포체 조직편을 분석하고 이 조직편들을 3D 컴퓨터 모델로 구현합니다.
고해상도의 2D 이미지 데이터를 분석한 후 3D로 재구성하여 등록하기 위해서는 고도의 데이터와 연산력이 요구되는데요. 율리히 연구소의JURON 슈퍼컴퓨터는 이 작업을 수행하기 위해 적합한 역량을 갖추고 있습니다. JURON 시스템은 엔비디아 테슬라(Tesla) P100 GPU 가속기를 탑재해 동시 등록 작업을 신속하게 처리할 수 있답니다.
딥 러닝, 3D 맵 작성을 가속화
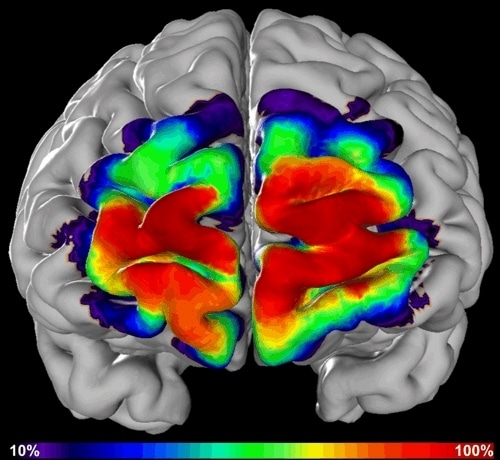
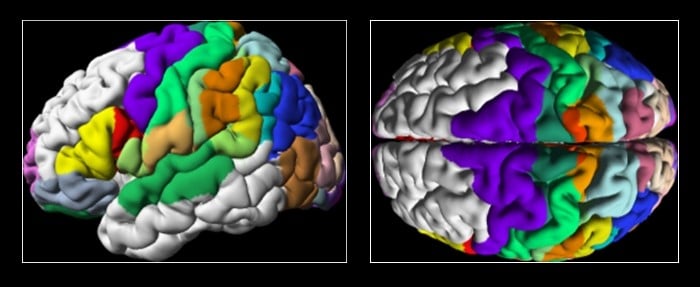
전통적으로 과학자들은 뇌의 3D 디지털 지도를 구축하기 위해 조직편의 조직 분포 유형을 분석해왔습니다. 세포체를 염색하면 뇌의 구획 별 경계를 파악할 수 있는데요. 여러 뇌를 대상으로 이 과정을 거치면 공통의 3D 레퍼런스 모델을 얻을 수 있습니다³. 뇌의 특정 영역이 구체적인 위치에서 발견될 확률을 나타내는 지도도 만들 수도 있지요. 현재까지는 인간 뇌의 약 70%에 달하는 200여개 구획이 밝혀져 있습니다.
하지만 이 방법은 시간이 많이 소모되고, 확장성이 없으므로 고출력 이미징이 어렵습니다. 그리하여 카트린 아문츠(Katrin Amunts) 교수와 티모 딕샤이드(Timo Dickscheid) 박사가 이끄는 율리히 연구진은 이 과정을 가속화시키기 위해 딥 러닝을 적용합니다.4 연구진은 고해상도의 세포체 염색 이미지로부터 얻은 텍스처 특징을 결합시키는 CNN(convolutional neural networks)을 트레이닝 했습니다.
연구진은 이 모델을 뇌의 시각계에 적용하여 제한된 부분 주석을 바탕으로 모델 트레이닝을 했습니다. 그리고 이를 통해 이전에는 보이지 않던 뇌 구역에서 공간적으로 일관성이 있고, 재현 가능한 예측을 하였습니다.
이와 같은 3D 뇌 모델은 건강한 뇌 구조와 기능에 대한 학계의 이해에 중대한 진전을 이루어 냅니다. 의료진은 이 모델에 기반한 뇌 지도 정보와 첨단 이미징 기법으로 추출한 환자 데이터를 비교해 볼 수 있습니다. 지도와 환자 데이터 상의 차이를 파악하면 파킨슨병, 알츠하이머, 뇌졸중 등 광범위한 신경계 질환 및 신경퇴행질환에 대해 유용한 정보를 얻을 수도 있습니다.
JURON 클러스터는 IBM과 엔비디아가 율리히 연구소에서 구현한 두 개의 파일럿 시스템 중 하나인데요. JURON 클러스터는 IBM 민스키(Minsky) 서버 18대로 구성되어 있으며, 엔비디아 NVLink 인터커넥션 기술에 기반한 테슬라(Tesla) P100 GPU 가속기가 서버당 4개씩 탑재되어 있습니다. JURON 시스템에 대한 보다 상세한 내용은 여기에서 확인하세요.
<참고문헌>
- 카트린 아문츠(Katrin Amunts), 크리스토프 이벨(Christoph Ebell), 제프 뮐러(Jeff Muller), 마틴 텔레폰트(Martin Telefont), 알로이스 놀(Alois Knoll), 토마스 리퍼트(Thomas Lippert) (2016): <인간 뇌 프로젝트: 인간 뇌를 독해하기 위한 유럽 연구 인프라(The Human Brain Project: Creating a European Research Infrastructure to Decode the Human Brain)>, 뉴런 92, 11. 2, 2016
- 카트린 아문츠 외Lepage, C., Borgeat, L., Mohlberg, H., Dickscheid, T., Rousseau, M., Bludau, S., Bazin, P., Lewis, L., Oros-Peusquens, A., Shah, N., Lippert, T., Zilles, K., Evans, A 2013 <빅 브레인(BigBrain) – 초고해상도 3D 인간 뇌 모델>, 사이언스 지 340호 6139 pp. 1472-1475
- 카트린 아문트, 질(Zilles) K. (2015) <브로드만을 넘어서: 인간 두뇌의 건축술 지도(Architectonic Mapping of the Human Brain beyond Brodmann.)> 뉴런 2015 12. 16;88(6):1086-107
- H. 스피처(Spitzer), K.), 카트린 아문트, S. 하멜링(Harmeling), T. Dickscheid (2017): <CNN을 이용한 고해상도 조직학 뇌 섹션에서의 시각적 피층 분획(Parcellation of Visual Cortex on high-resolution histological Brain Sections using Convolutional Neural Networks)>, IEEE 국제 생물 의학 심포지엄 생체의학 이미지(ISBI)