재택근무 중인 에이다 세도바(Ada Sedova)는 가끔은 잠옷 차림으로 신종 코로나바이러스 감염증(코로나19) 감염을 막을 수 있는 미세분자를 찾기 위해 세상에서 가장 강력한 슈퍼컴퓨터를 사용합니다.
오크리지 국립연구소(Oak Ridge National Laboratory)의 생물물리학 연구원인 세도바는 “지금 그 어느때보다 바쁘게 일하고 있습니다. 코로나19 팬데믹에 대한 불안감이 매우 높은 지금 저도 굉장히 많은 개인 시간을 그 해결책을 찾는데 보내고 있습니다”라고 말했습니다.
이런 노력으로 언젠가 수십억 달러의 수익을 창출하는 날이 올 수도 있겠죠? 단 하루만에 20억개의 분자 테스트를 하는 날도 있다는 데요.
세도바는 수십 개의 원자보다도 크기가 작은 유기 분자인 리간드(ligand)를 찾고 있습니다. 리간드를 제대로만 찾으면 코로나바이러스의 단백질에 딱 붙어 바이러스가 건강한 세포를 감염시키는 것을 막을 겁니다.
그러나 문제는 확인해야 하는 리간드와 단백질 수가 너무 많은 데다가 내포하고 있는 원자력이 변화할 때마다 리간드와 단백질의 형태도 같이 바뀐다는 겁니다. 이를 일일이 확인하는 작업은 수십억 개의 화합물로 이뤄진 사막에서 너무나도 작은 바늘을 찾는 것과 마찬가지입니다.
습식 실험실의 전문가들이 그런 가능성을 하나 하나씩 분석하려면 수 년이 걸릴 겁니다. 오크리지 국립연구소의 슈퍼컴퓨터 ‘서밋(Summit)’에서 9,216대의 CPU로 모든 가능성을 시뮬레이션해도 4년이 걸릴 수 있습니다. 이런 상황이다 보니 세도바 연구팀이 연구 속도를 높이기 위해 서밋에서 구동되는 27,648 대의 엔비디아 GPU로 눈을 돌리게 된 것이죠.
세도바의 연구팀은 독일의 다름슈타트 공과대학(TU Darmstadt)과 협력해 스크립스 연구소(Scripps Research)에서 개발해 단백질과 리간드가 결합하는 방식을 시뮬레이션 하는 오픈소스 프로그램인 오토덕(AutoDock)의 OpenCL 버전을 사용하기 시작했습니다.
엔비디아 쿠다로 구현한 엄청난 속도
엔비디아와 스크립스 연구소의 도움으로 연구팀은 쿠다(CUDA)가 서밋에서 실행되도록 코드를 포팅해 처리속도를 2.8배 가속화했습니다. 미국 소프트웨어 컨설팅 기업인 쥬빌리 디벨롭먼트(Jubilee Development)의 애론 샤인버그(Aaron Scheinberg) 연구원은 데이터가 GPU로 피드되는 속도를 가속화하기 위해 OpenMp를 사용할 수 있는 방법을 찾고 작업 속도를 추가로 3배 높였습니다.
세도바는 또 다른 테스트에서 단백질에서 14억개의 화합물의 데이터 세트를 단 12시간 만에 높은 정확도로 스크리닝할 수 있다는 것을 입증했습니다. 이것은 CPU 프로그램 대비 33배 이상 빠른 속도입니다.
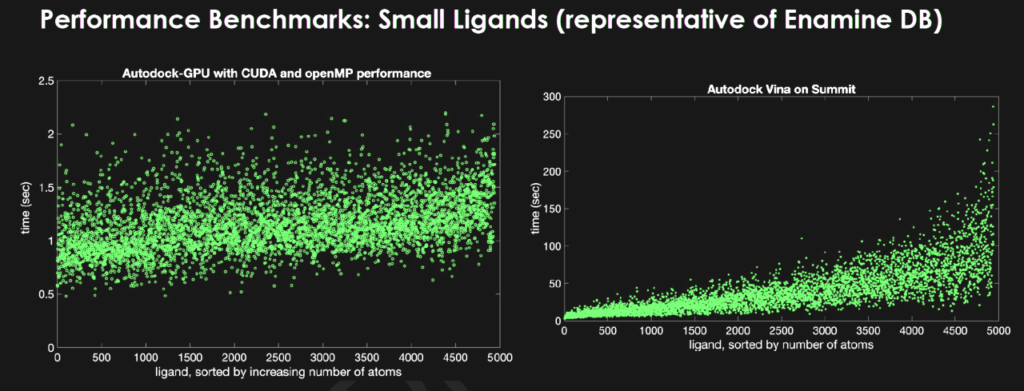
세도바는 “GPU가 서밋의 크기, 아키텍처와 만나 이전 대비 수십억 개나 더 많은 화합물을 결합할 수 있게 됐습니다”라고 말했습니다.
이 연구팀의 또 다른 멤버인 생물물리학자 조쉬 버마스(Josh Vermaas)는 오토덕을 쿠다로 포팅하는 작업을 돕는 엔비디아의 스캇 르 그란드(Scott Le Grand)에게 감사를 표하며 작업 과정을 설명한 블로그에 “스캇 르 그란드는 과거 OpenCL 전용 코드로만 사용되던 성능을 크게 향상시키는 데 엄청난 도움을 줬습니다”라고 말했습니다.
하루만에 끝낸 20억개 화합물 시뮬레이션
세도바는 앞으로 시뮬레이션 성능이 24시간만에 무려 20억개의 화합물을 분석할 수 있을 정도로 대폭 향상될 것이라고 기대하고 있습니다. 성공한다면 이런 규모의 고해상도 시뮬레이션의 최초 사례가 될 것입니다.
그러나 그 전에 해결해야 할 몇 가지 걸림돌이 남아있는데요.
단백질과 리간드의 도킹(docking, 결합)을 위한 표준 워크플로우는 저속 파일에 기반한 프로세스로 진행됩니다. 노트북에서 수백 가지의 화합물을 테스트하는 경우에는 문제가 되지 않겠지만 수십만 건 규모의 파일을 테스트하는 것이라면 세계에서 가장 큰 슈퍼컴퓨터에서 처리한다 해도 속도가 저하될 수 있습니다.
그래서 이와 같은 속도 저하 문제를 해결하는데 관심 있는 오픈소스 과학자들의 적극적인 도움이 필요합니다.
세도바 연구팀은 서밋에서 대량 작업을 안전하게 착수할 수 있도록 보장하는 새로운 워크플로우를 만들어 가면서 가속 컴퓨팅 분야를 선도하고 있습니다. 세도바는 모든 리간드의 정보를 담은 데이터베이스를 구축하기 위해 시스템 I/O 전문가들과 논의 중입니다.
그 다음엔 서밋의 4,608 노드 중 108개를 사용해 약 100만개의 화합물에 대한 실험을 시작하려고 하고 있습니다. 세도바는 “실제로 계획대로 진행이 된다면 서밋의 모든 노드를 활용해 140억개의 화합물을 실험하는 프로젝트를 진행할 예정입니다”라고 밝혔습니다.
유망 분자 선별 작업
세도바 연구팀이 성공할 경우 가장 성공 가능성이 높은 화합물 약 9,000개를 멤피스(Memphis) 연구진에게 보내 이를 습식 실험실에서 실제 바이러스로 테스트하게 되는데요. 그렇게 되면 더 이상 사막에서 바늘 찾기가 아니라, 조그마한 잡초 더미에서 바늘 찾기가 되겠죠.
이 모든 것은 오크리지 국립연구소 수석 연구원 제레미 C. 스미스(Jeremy C. Smith)가 지난 1월 코로나19 퇴치 약품 연구를 위해 서밋을 사용하면서 처음 시작됐습니다. 물론 아직 초기 단계에 머물러 있긴 합니다.
세도바는 앞으로 단백질-리간드 결합 분야를 고성능 컴퓨팅에서 일반적으로 사용되는 방법과 연계할 수 있는 다른 방법을 찾아볼 생각입니다. 아직도 이 모든 일을 할 에너지가 넘친다고 하네요.
엔비디아 코로나19 페이지를 클릭해 코로나바이러스 퇴치를 위해 GPU를 활용한 다양한 사례들을 살펴보세요.
